因為我發現很難將人類基因組的結構這樣微小的東西聯絡起來,所以我喜歡把它放大一百萬倍來想象。在這個尺寸下,每個DNA分子——一條染色體——都像拉麵條一樣寬。如果將構成細胞基因組的全部46條放大後的染色體首尾相連,它們將從紐約延伸到堪薩斯城,儘管它們實際上是摺疊起來以適應房屋大小的結構——細胞核內部。總共有46條染色體,包含大約兩組20,000個基因。每個基因都拼寫出一個編碼資訊,告訴細胞如何製造特定的蛋白質;在百萬倍的尺度下,一個基因和一輛汽車一樣長。
窺視細胞核內部,你會看到DNA在不停地擺動。大約十年前,當我還是博士生的時候,我一邊攪拌晚餐裡的拉麵條,一邊想知道基因組是如何避免像我的麵條一樣纏結成一團糟,從而阻止其關鍵的遺傳資訊被髮送出去的。
2014年,我和我的同事為這個問題的一個答案做出了貢獻,進一步認識到細胞核內部基因組的結構遠非隨機。我們位於貝勒醫學院的團隊,由我的學生Suhas Rao、Miriam Huntley和Adrian Sanborn領導,發現人類基因組以形成約10,000個環的方式摺疊。這些環遵循一個簡單的程式碼,隱藏在基因組本身的序列中。事實證明,它們是古老的結構;許多相同的環也出現在小鼠中,這是6000多萬年前的祖先物種的共同遺產。這種跨越時間的永續性表明,這些環對生存至關重要。
支援科學新聞報道
如果您喜歡這篇文章,請考慮透過以下方式支援我們屢獲殊榮的新聞報道 訂閱。透過購買訂閱,您正在幫助確保有關塑造我們當今世界的發現和想法的具有影響力的故事的未來。
這些環似乎有助於控制基因活動。所有細胞都具有相同的基因,但是如果活動模式沒有差異,身體就不可能存在:心肌細胞與腦細胞將沒有區別。這些獨特的模式是如何協調的,一直是一個謎。現在看來,環是模式控制器之一,是基因管絃樂隊的指揮,影響著特定基因何時變得足夠活躍以影響細胞功能。
隨著我們繼續探索這些環,我們期望更好地理解基因調控,並找到許多疾病如何產生的線索。最近,我們和其他人已經弄清楚了這些環是如何形成的,跳著優雅的探戈,使基因組保持無纏結狀態。
基因組的Facebook
我對DNA纏結的思考與一個更大的問題有關:細胞核中DNA的3-D排列如何影響基因活動?自1970年代後期以來,越來越多的證據表明,啟用基因需要稱為增強子的DNA小片段。生物學家還了解到,增強子可以位於DNA鏈中遠離其靶基因的位置。為了觸發基因的“開啟”開關——基因附近的DNA片段,稱為啟動子——該鏈大概必須自身環回,使增強子靠近啟動子。但是這種假設正確嗎?我被這個問題迷住了,只能想到一種解決它的方法:找到所有環。
從概念上講,這樣做的計劃很簡單。如果兩個人特別頻繁地在一起,那麼可以邏輯地推斷他們是朋友。同樣,我們推斷,如果染色體上相距遙遠的兩段DNA(“位點”)傾向於特別頻繁地在一起,那麼DNA可能已經摺疊成一個環。我們需要的是一種方法來測量基因組的各個部分相互作用的頻率:為人類基因組構建類似Facebook的東西。
為了將我們的想法變為現實,我們採用了1993年當時在範德比爾特大學的Katherine Cullen及其同事描述的一種方法。當時,基因組使所有已知的成像形式都感到困惑:就像一個糟糕的肖像物件一樣,擺動的染色體面條拒絕靜止不動。但是,庫倫利用了這種不穩定的染色體來發揮她的優勢。她知道,隨著染色體的晃動,基因組的不同部分會相互碰撞。在3-D中非常接近的部分會相互碰撞很多次;相距遙遠的部分只會偶爾接觸。因此,如果您可以測量碰撞頻率,則可以弄清楚基因組的哪些部分在3-D空間中彼此靠近。
為了測量這種碰撞頻率,庫倫和她的同事開發了他們稱之為核連線測定(NLA)的方法。本質上,您取出細胞,並在不破壞其細胞核的情況下,穩定其基因組。然後,您送入一種酶將DNA切成小片段,並部署一種蛋白質,將兩個附近片段的末端融合,形成單鏈。最後,您檢查融合片段集合中DNA鹼基對(DNA程式碼的配對字母,形成DNA熟悉的“梯子”的“梯級”)的序列。如果在細胞接細胞中,您看到特定DNA片段對的融合,而這些片段最初並非在染色體上彼此相鄰(稱為連線連線點),則可以得出結論,這兩個DNA片段經常在細胞核的3-D空間中彼此靠近。
庫倫在科學雜誌上發表的見解使她能夠證明,DNA特定長片段兩端的兩個DNA片段比偶然預測的頻率更高地相互碰撞。換句話說,DNA形成了一個環。
早在1993年,使用核連線測定進行的實驗很難執行。幸運的是,當我在2000年代中期作為研究生看到庫倫的論文時,已經有了一個可用的參考人類基因組,並且DNA測序變得非常便宜。我和麻省理工學院和哈佛大學布羅德研究所的其他三個人——Chad Nusbaum、Andreas Gnirke和Eric Lander——勾勒出一種方法,該方法將分析的接觸頻率不是單個DNA位置對的接觸頻率,而是整個基因組中每個位置對的接觸頻率。它還將使我們能夠精確地查明每個連線連線點的一半來自何處。
我們決定將我們的新方法建立在馬薩諸塞大學醫學院的Job Dekker開發的庫倫程式變體之上。與庫倫使用完整細胞核不同,德克爾炸開了細胞核,並在極其稀釋的溶液中執行了關鍵的連線步驟。德克爾普及並稱為“染色體構象捕獲”或“3C”的這種修改,被認為可以產生更可靠的碰撞頻率估計值。
接下來,我們在3C中添加了一些步驟。在將片段粘合在一起之前,我們將在破碎的DNA末端附上易於檢測的標籤——以標記兩個附近片段連線的位置。完成此步驟後,我們將把粘合的片段切成更小的片段,並僅拉出帶有標籤的片段;這些片段將包含純連線連線點。與德克爾、他當時的博士後研究員Nynke van Berkum和布羅德研究所的Louise Williams合作,我們發現我們可以一次識別數百萬個接觸。我將該方法稱為“Hi-C”,這是對“3C”和我小時候最喜歡的飲料之一的名稱的模仿。我們在2009年發表了該方法。
我們最早的全基因組Hi-C圖譜顯示,染色體儘管不停地擺動,但並沒有在細胞核內隨機混亂地摺疊起來。相反,每條染色體都被劃分為域:包含彼此頻繁接觸的片段的DNA片段。一個域中的位點與另一個域中的位點相互作用的頻率較低。更重要的是,我們的Hi-C資料揭示,每個域都位於細胞核中兩個較大的空間鄰域之一內。我們將這些鄰域稱為“區室”,並標記為A和B。
我們發現A區室富含遺傳活動標記,例如信使RNA,信使RNA是基因傳送出去以告知細胞其餘部分該做什麼的分子。B區室更密集,並且基本上處於非活動狀態。當域開啟或關閉時,它們會從一個區室移動到另一個區室。(今天我們知道細胞核包含多個A和B亞區室。)
這種動態區室化的發現使我們感到興奮,因為它證實了基因組的大規模3-D結構並非隨機的,而是與基因活動密切相關的。但是,我感到失望的是,Hi-C資料中似乎從未出現過一種摺疊特徵:環!
Hi-C資料通常表示為熱圖:一個圖,顯示染色體中兩個位點相互形成接觸的頻率。在這樣的圖中,兩個位點之間的接觸頻率由x軸和y軸上點的亮度表示,代表位點的交點。環應該表現為一個異常明亮的點,對應於環的兩個錨點。但是我們沒有看到任何這樣的亮度峰值。如果我們不能證明環正在形成,我們就無法探索增強子是否透過物理上靠近啟動子來啟用基因。
製作環圖
這個問題困擾了我們三年。然後,在2012年,Rao和Huntley弄清楚了哪裡出了問題。他們意識到Hi-C的一個方面——在連線之前破壞細胞核——破壞了環等精細結構。因此,他們著手開發一種更新的Hi-C方法,該方法在連線過程中保持細胞核完整。
這種稱為原位Hi-C的新方法產生了巨大的不同。在對白細胞的研究中,Rao和Huntley發現,明亮的峰值現在出現在我們熱圖的各個位置,每個峰值代表一個推定的環。但是,自從我開始致力於繪製環圖以來,已經過去了六年;我不再相信自己的眼睛。我的團隊和我擔心我們可能在資料中看到了實際上不存在的東西。
為了確保我沒有受到確認偏差的影響,我把地圖帶回家給我的兒子加布裡埃爾,當時他三歲。“你看到一個紅點嗎?”“是的,”他說。“你能指出來嗎?”他可以。
我們成功了:一張顯示10,000個環的地圖,分佈在整個人類基因組中。我們檢查了這些環是否連線了基因啟動子和增強子。它們經常這樣做。
在進一步的測試中,我們將我們的血細胞圖譜與另一種細胞——來自肺部的細胞的新圖譜進行了比較。我們看到了許多相同的環,但我們也看到了我們推測涉及不同增強子和不同靶基因的新連線。環模式的這些變化表明,環可能參與調節賦予細胞獨特身份的基因。
我們想知道環化是否是人類獨有的,或者其他生物體中是否存在相同的環。因此,我們繪製了小鼠細胞中環的圖譜,發現一半的環存在於人類基因組的相應位置。這些共享的環在至少6000萬年的進化過程中得以儲存,從科羅拉多河開始雕刻大峽谷之前很久就在地球上漫遊的祖先生物開始。
環的邏輯
我們資料的一個有趣的含義是,環不是靜態的:它們似乎不斷產生、分離並再次形成。自然,我們想知道這是如何工作的。
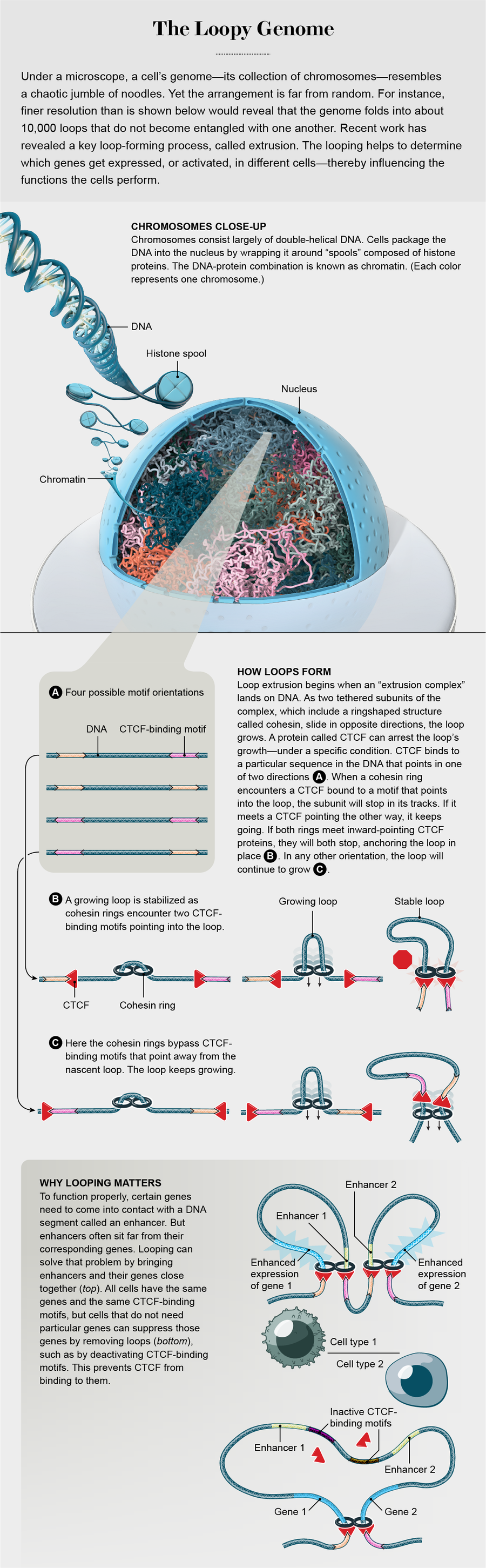
我們懷疑數百種蛋白質參與其中。然而,資料講述了一個不同的故事。在一個又一個的環中,突出了兩個蛋白質因子。一個名為CTCF,由Victor Lobanenkov及其同事於1990年發現。它包含11個稱為鋅指的成分,使CTCF能夠非常緊密地結合到DNA上的特定位點。第二個因子,粘連蛋白,由現任牛津大學的Kim Nasmyth於1997年發現,是一個由多種蛋白質組成的環狀複合物。人們認為,兩個粘連蛋白環可能會連線起來並協同工作,這對環中的每個環都環繞DNA並在其上自由滑動,就像項鍊上的環一樣。
圖片來源:Falconieri Visuals
看到這些蛋白質並不完全令人驚訝:許多早期的研究表明它們可能參與基因組摺疊,儘管在環錨點(尤其是在連線啟動子和增強子的環處)起如此普遍的作用是出乎意料的。
然後我們偶然發現了一些真正奇怪的東西。Rao、Huntley和我要求實驗室的一位新的計算科學家Ido Machol研究組蛋白(有助於將DNA包裝在細胞核內部)在CTCF分子附近的分佈。Machol注意到,環外側的組蛋白比內側的組蛋白更多,就好像組蛋白以某種方式知道環相對於CTCF分子的位置一樣。我懷疑這個發現只是反映了Machol程式碼中的一個錯誤。但是隨著時間的推移,Machol沒有發現任何錯誤。
我們開始尋找生物學解釋。在描述CTCF發現的原始論文中,Lobanenkov表明CTCF並非附著在DNA上的任意位置。相反,它總是結合到特定的DNA詞——大約20個鹼基的特定序列,稱為基序。由於DNA是雙螺旋結構,因此它具有兩條鏈。基序可以出現在任一條鏈上,指向巨大的DNA麵條的任一端。DNA基序的相對方向通常是隨機的,就像拋硬幣一樣:典型的基序有50%的機率指向一個末端,而有50%的機率指向另一個末端。因此,起初,我們希望看到環錨點處CTCF結合基序的隨機方向。
我們想知道環錨點處的CTCF結合基序是否給組蛋白提供了線索,告訴它們應該在哪裡連線到基序附近的DNA。我們進行了檢查,令我們驚訝的是,即使兩個微小的CTCF結合基序在未摺疊的DNA中相隔數百萬個DNA字母,它們也總是彼此指向,並指向環內,我們將其命名為會聚方向。這種會聚規則解釋了組蛋白如何知道自身定位的位置——它們只需要確定CTCF結合基序的指向方向即可。
但是,在解決一個難題的同時,會聚規則又製造了第二個更大的謎團。基序的非隨機方向與預期相反。為了便於理解,讓我們再次將基因組放大一百萬倍。現在,基序的長度均為五毫米,並且被長達一公里的基因組麵條分隔開。然而,不知何故,彷彿受到神奇指南針的引導,形成環狀DNA片段兩端的基序總是彼此指向。像任何好的魔術技巧一樣,會聚規則似乎在物理上是不可能的。這也與人們普遍接受的環可能形成的方式相矛盾。
當時,幾乎所有人(包括我們自己)都認為基因組環是透過擴散形成的。在該方案中,形成環所需的蛋白質在DNA片段的一端結合。接下來,另一個環使能蛋白在另一端結合。然後,像往常一樣,DNA擺動。最後,如果擺動使兩種蛋白質結合在一起,它們就會形成物理連線,從而形成一個環。問題是,整個DNA鏈有太多的擺動空間,如果擴散模型是正確的,那麼CTCF結合基序的相對方向就無關緊要了。然而,我們卻看到了會聚。在一年之內,由倫敦大學學院的Suzana Hadjur領導的一個團隊和由傑克遜實驗室的Yijun Ruan領導的一個團隊在他們自己的資料集證實了會聚規則。該規則將繼續存在,我們看到的環不可能透過擴散形成。
驅動式環化
如果環不是透過擴散形成的,那麼它們是如何產生的?CTCF和粘連蛋白的作用是什麼?在基因組摺疊緊急情況下,我們總是會這樣做:我們開始擺弄耳機線。
我非常確定,大多數從事基因組摺疊工作的人都會隨身攜帶一個長長的麵條狀物體:一根紗線,一根塑膠管。當您遇到難題時,您會拿出這個物體並擺弄一番。有一天,當我們探索環形成的可能模型時,Rao和我來回傳遞耳機。突然,我們意識到答案不在我們的耳機中;而是在我們的揹包上。
想象一下調節揹包帶長度的裝置。這個物體稱為三滑扣,或多或少由兩個彼此物理連線的環組成。帶子從第一個環進入,然後從第二個環出來。如果您想調整帶子長度,則可以將一些帶子拉過其中一個環並開始形成一個環。您可以繼續擴大環的尺寸,直到到達一塊摺疊的材料阻止您。
也許成對的粘連蛋白環像三滑扣一樣工作?起初,它們附著在基因組上的任何位置,DNA進入一個環並從另一個環出來。但是隨後,這兩個環沿相反的方向滑動(一個沿線性分子向左滑動,一個向右滑動),在滑動過程中擠出一個不斷增長的環。但是它們不會永遠滑動。最終,一個環接近CTCF分子結合的位點。如果底層的CTCF結合基序指向接近的環,則滑動環會在接觸時停止。但是,如果基序朝另一個方向,則粘連蛋白會忽略它並繼續前進。(透過這種方式,CTCF結合基序就像粘連蛋白交通的停車標誌:如果標誌朝向您,則您停止;如果標誌朝向另一個方向,則您不會停止。)第二個環繼續前進,直到它也到達一個朝內指向的CTCF結合基序。現在環已完成。
如果粘連蛋白環實際上以這種方式工作,那麼環將僅在符合會聚規則的CTCF結合基序對之間形成。我們很快意識到,這種擠出過程將為細胞提供至關重要的好處。如果環透過擴散形成,那麼染色體中的環對很容易纏繞在一起,導致染色體形成結並相互纏結。這將使基因難以正常運作,並可能阻止染色體在細胞需要分裂時分離。相反,透過擠出產生的環不會形成結或纏結——這就是為什麼無論您用三滑扣調整揹包帶的長度多少,揹包帶都不會打結的原因。
該模型是瘋狂的猜測。它做出了許多我們沒有直接證據的基本假設,例如粘連蛋白可以沿DNA滑動的概念。我們擔心我們瘋了。但是,當我們閱讀有關粘連蛋白的文獻時,我們意識到Nasmyth本人早在2001年就提出粘連蛋白可能會擠出DNA。Sanborn運行了詳細的模擬,這些模擬密切概括了我們地圖中的資料。當Rao在真實DNA上進行實驗時,環化變化的方式與Sanborn的模型預測的完全相同。
刪除環錨點處的CTCF結合基序消除了環。翻轉基序的方向會使原始環消失,但會在另一側形成另一個環。新增CTCF結合基序——只要它指向正確的方向——也會導致新環的形成。然後我們發現我們可以隨意地向基因組新增和刪除環。
我們迅速撰寫並提交了一篇關於我們的擠出模型和我們為測試該模型而進行的環工程實驗的論文。該領域正在升溫,在2015年末的幾個星期內,我們的實驗室和其他兩個團隊發表了論文,證明了這種3-D基因組手術是有效的。同樣,三個團隊——我們的團隊、埃默裡大學的一個團隊和麻省理工學院的一個團隊——報告說,會聚規則支援環透過擠出形成的模型。最終,科學界開始解開環的邏輯。
進展仍在繼續,現在正以驚人的速度進行。在格萊斯頓研究所,Benoit Bruneau及其同事表明,干擾CTCF會大大削弱環。在歐洲分子生物學實驗室,Francois Spitz及其同事透過消除一種被認為將粘連蛋白載入到DNA上的蛋白質而獲得了類似的結果。在荷蘭癌症研究所,Benjamin Rowland的團隊表明,消除一種從DNA中去除粘連蛋白的因子會導致更大的環,這可能是因為粘連蛋白現在可以滑動更長的時間。在我們實驗室,Rao表明,透過降解粘連蛋白本身,我們可以在幾分鐘內消除所有粘連蛋白環。
但是我們都渴望得到直接的證實:親眼目睹擠出作用。最終,在2018年4月,荷蘭代爾夫特理工大學的Cees Dekker及其同事做到了這一點。透過使用酵母的縮合蛋白——一種與粘連蛋白密切相關的蛋白質複合物——他們製作了一部顯微電影,核結構領域的許多人將永遠不會忘記。首先,您會看到一條DNA帶。然後,縮合蛋白著陸,形成一個小的DNA結節。結節不斷生長,直到觀看者意識到它的真實含義:一個擠出的環。
轉向健康
隨著環形成的機制和規則的出現,環化對於健康和疾病的重要性變得越來越清晰。例如,哈佛大學的Frederick Alt及其同事已經開始闡明環化在抗體產生中起的作用。您的身體透過剪下和貼上抗體基因片段來製造以前從未遇到過的病原體的抗體。Alt的團隊發現,這個過程是透過形成多個CTCF錨定的環,然後將它們切除來完成的。
柏林馬克斯·普朗克分子遺傳學研究所的Stefan Mundlos實驗室表明,修飾小鼠中的單個CTCF結合基序會導致動物的爪子發育出異常數量的腳趾。具有相應變化的人類沒有五根手指。美國國立衛生研究院的Rafael Casellas表明,破壞小鼠漿細胞瘤(一種癌症)中的CTCF結合基序可以使腫瘤的生長速度減慢40%。
然而,隨著環擠出概念越來越受到重視,關於環在基因調控中起作用的更深層理論開始瓦解。幾十年來,科學家們認為環像開關一樣工作:當增強子和啟動子之間的環存在時,相應的基因就會開啟。因此,我們預計,當我們從細胞中去除粘連蛋白時,基因表達會變得混亂,成千上萬的基因會改變其活性水平。正如預測的那樣,許多基因確實發生了變化。但是變化相當小。環——至少是擠出形成的環——畢竟不是二元開關。相反,它們似乎更像旋鈕,將基因活性向上或向下稍微調整一點,微調細胞的不同蛋白質的供應。
換句話說,大自然讓我們陷入了一個迴圈。我們以為我們理解了遊戲規則,環會開啟基因。但是現在我們已經看到了環的實際作用,我們必須承認我們的視野過於簡單了。甚至有可能基因調控可能只是環的副業;也許它們在細胞中的主要功能完全是另一回事。
像任何在未開發領域探險的探險家一樣,我們需要更好的地圖。我在美國國立衛生研究院的DNA元件百科全書(ENCODE)專案的同事Ruan和我目前正在與我們的同事合作,建立人類基因組中環化的第一個圖譜,繪製整個人體組織中的環。我們的團隊和許多其他團隊也加入了4D核組聯盟,該聯盟正在開發解決這些問題的新方法。我的實驗室的博士後Olga Dudchenko建立了DNA動物園——一個由世界各地的學術實驗室、動物園和水族館組成的聯盟,該聯盟正試圖組裝數百個物種的基因組,記錄生命之樹中環的進化。
對於研究人員來說,一個科學故事的結尾始終是另一個科學故事的開始。二十億年前,在細胞核出現之前,DNA擠出過程就出現了。為什麼?再次,進入迴圈。

